Искусственный интеллект для физики белка
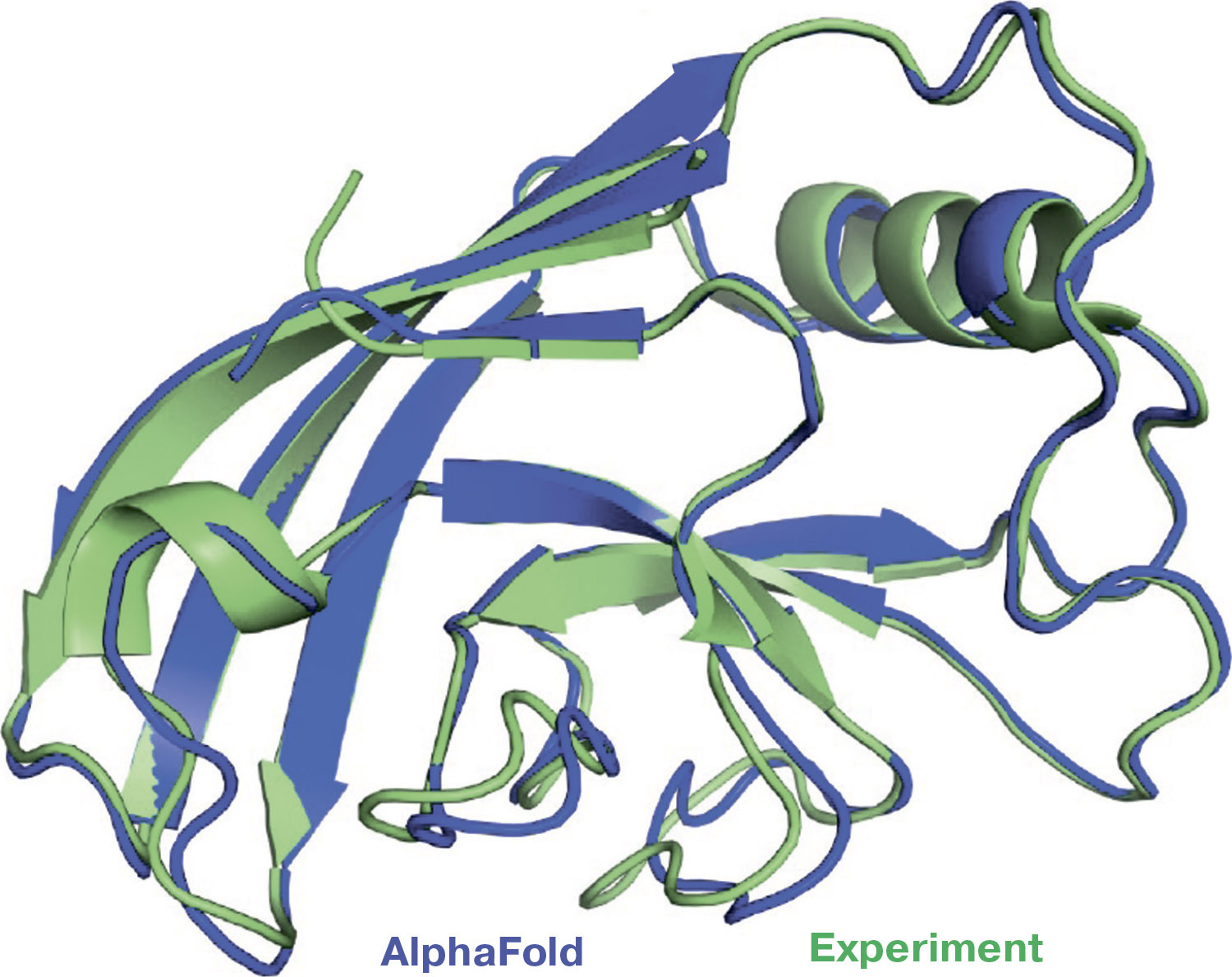
Не так давно мировая наука получила в своё распоряжение новую разновидность искусственного интеллекта — программу AlphaFold. Порой можно услышать, что эта программа заменяет собой целую область молекулярной биологии — ту, где занимаются пространственными структурами белков. AlphaFold действительно использует весьма и весьма успешный алгоритм в предсказании трёхмерных «портретов» белковых молекул. Однако прежде чем говорить о революционном успехе, нужно уточнить, что именно предсказывает искусственный интеллект этой программы, как он это делает и что он «знает».
В чем сила программы AlphaFold
Белки — это и молекулярные машины, и строительные блоки, и оружие живой клетки. Белки образованы полипептидными цепями, обычно весьма длинными, включающими сотни аминокислотных остатков. Клетка синтезирует белки из двадцати основных аминокислот, последовательность которых в полипептидных цепях кодируется генами. Но ни один белок не существует в виде прямой «палки» из сшитых друг с другом аминокислот. Аминокислотные остатки полипептидной цепи неизбежно взаимодействуют между собой. Физико-химические взаимодействия определяют геометрию связей между атомами, их сближение и отталкивание, их отношения с окружающей средой — водой, ионами, другими молекулами. Полипептидная цепь многократно изгибается, накладывается сама на себя — будучи изначально неупорядоченной, она приобретает определённую, трёхмерную структуру. Происходит то, что называется самоорганизацией, или сворачиванием белка. Именно в свёрнутом виде белок, можно сказать, становится самим собой. Биологические функции белков тесно связаны с тем, как выглядят их трёхмерные (3D) структуры.
Многие тысячи таких пространственных структур уже определены экспериментально — с помощью рентгеноструктурного анализа, ядерного магнитного резонанса и криоэлектронной микроскопии. Эти трёхмерные «портреты» белков хранятся — в виде огромных наборов 3D-координат всех атомов белка — в компьютерном банке белковых данных PDB (Protein Data Bank) и других подобных банках. Однако рентгеноструктурный анализ, ядерный магнитный резонанс и криоэлектронная микроскопия весьма трудоёмки. Поэтому миллиарды белков из самых разных организмов, от вирусов и бактерий до позвоночных, до сих пор остаются с неизвестными пространственными структурами. А их полезно знать — в частности (но не только), для создания новых лекарств.
В то же время для сотен миллионов белков уже известны аминокислотные последовательности их полипептидных цепей, определять которые много легче. Но их пространственная структура остаётся тайной.
Поэтому большой интерес вызывают методы предсказания белковых 3D-структур по аминокислотным последовательностям. Как уже было сказано, сворачивание белка происходит благодаря взаимодействию аминокислотных остатков друг с другом, а также и с кофакторами (гемами или другими макромолекулами), и с окружающей средой. Необходимо подчеркнуть, что пространственная структура — результат самоорганизации, то есть структуры белковых цепей определяются самими аминокислотными последовательностями этих цепей (по крайней мере, если речь о глобулярных белках). Напрашивается вывод, что по аминокислотным последовательностям можно предсказать, в какую 3D-форму свернётся полипептидная цепь. Здесь, однако, нужно уточнить, что сворачивание (самоорганизацию) белка можно понимать двояко — как процесс и как результат.
У «средней» белковой цепи есть огромное количество вариантов пространственной структуры, порядка 10100. Перебор всех 10100 альтернатив потребовал бы миллиардов миллиардов лет. Между тем белок сворачивается в одну и ту же уникальную, свойственную ему трёхмерную структуру всего за несколько секунд или минут1. Решая проблему сворачивания в смысле процесса (то, что порой называют «проблемой сворачивания белка первого рода»), мы отвечаем на вопрос, как может белковая цепь чрезвычайно быстро выбрать свою структуру из гигантского набора возможных вариантов.
1 Почти мгновенный выбор единственной нужной структуры из колоссального количества возможных называется парадоксом Левинталя, по имени молекулярного биолога Сайруса Левинталя, который сформулировал его в 1968 году. На решение этого парадокса потребовалось почти 30 лет.
Если же мы решаем проблему сворачивания в смысле результата (так называемая проблема сворачивания белка второго рода), то отвечаем на вопрос, какую именно структуру приобретёт белковая цепь с конкретной аминокислотной последовательностью.
Долгое время обе проблемы рассматривались как одна: предполагалось, что как только будет ясно «как может», сразу же станет ясно и «какую именно». Однако впоследствии выяснилось, что это две разные проблемы и решаются они по-разному. Поэтому мы можем позволить себе сейчас забыть о «как может» (тем более, что проблема «как может» решена2, и решена она была в нашем Институте белка РАН) и сосредоточиться на втором вопросе — какую пространственную структуру имеет белковая цепь с определённой аминокислотной последовательностью.
2Финкельштейн А. В., Бадретдинов А. Я. Физические причины быстрой самоорганизации стабильной пространственной структуры белков: Решение парадокса Левинталя. Мол. биол., 31, 469—477 (1997).
Интерес к пространственным структурам белков привёл к начавшимся в 1970-х годах своеобразным «соревнованиям» методов предсказания. С 1994 года такие «соревнования» проводились каждые два года в формате конференций по критической оценке структурных предсказаний, CASP (Critical Assessment of protein Structure Prediction). В этих конференциях участвовали сотни представителей десятков и сотен научных групп из множества стран. Методы предсказаний, использованные в CASP, первоначально базировались в основном на физике белковых цепей и на статистическом анализе уже известных белковых структур и контактов аминокислотных остатков в них.
Качество работы методов предсказания пространственных структур белков повышалось, но медленно, пока в 2018 году не появилась сделанная в Google DeepMind программа AlphaFold, а затем — AlphaFold 23. Эти программы были основаны на «глубоком обучении» многослойных нейронных сетей, и они сразу на голову — а AlphaFold 2 на две головы — превзошли все остальные «предсказывающие» программы.
3 Senior et al., Proteins 87, 1141—1148 (2019); Jumper et al., Nature 596, 583—589 (2021).
Яркий успех программы AlphaFold (а затем — и подобных ей) в определении трёхмерных белковых структур очевиден всем, кто работает в этой области науки, но остаётся ряд вопросов. Во-первых, в чём главная причина такого успеха?
Во-вторых, чем именно занимается программа AlphaFold? Основаны ли её предсказания 3D-структур на физике полипептидных цепей? Или алгоритм распознаёт 3D-структуры по сходству аминокислотных последовательностей разных цепей друг с другом — тех, для которых 3D-структуры неизвестны, с теми, для которых 3D-структуры уже получены? В-третьих, если структуры получаются из сходства аминокислотных цепей, можно ли с помощью AlphaFold извлекать из них какие-то физические закономерности? И нужны ли в принципе эти закономерности для успешных предсказаний?
Аминокислотные последовательности и структурные сходства
Если трёхмерная структура определяется аминокислотной последовательностью, то сам собой напрашивается вывод, что чем более схожи аминокислотные последовательности двух белков, тем более схожи они будут своими 3D-«портретами». Насколько должны быть похожи аминокислотные цепи для того, чтобы между ними возникла структурная схожесть? Возьмём два полипептида и вытянем их в линию рядом друг с другом, чтобы напротив первой аминокислоты одной цепи стояла первая аминокислота другой цепи. И вот, например, мы видим, что в двадцатом положении в обеих цепях стоит одна и та же аминокислота (скажем, аланин). А в двадцать первом положении аминокислоты разные. С двадцать второй по двадцать четвёртую позицию мы снова видим в обеих цепях одинаковые аминокислоты, например пролин-глицин-серин. Далее мы особого сходства не видим, но зато фрагмент с 30-й по 40-ю аминокислоты первой цепи очень похож на фрагмент с 38-й по 48-ю аминокислоты второй цепи... И так далее.
Такое совмещение полипептидных цепей с поиском сходств и различий в аминокислотной последовательности называется выравниванием. При выравнивании можно сдвигать цепи друг относительно друга и можно в одной цепи пренебречь каким-то участком последовательности, чтобы другой участок лучше совпал по аминокислотам со второй цепью. В таких случаях говорят про выравнивание со сдвигами, вставками и делециями, то есть исключениями небольших фрагментов аминокислотной последовательности. Это кажется подгонкой под ответ, однако на самом деле все такие сдвиги, делеции и вставки ясно видны при сравнении цепей родственных белков — например, глобинов разных животных, так что подобные манипуляции имитируют происходящие в живых организмах мутации и нередко позволяют хоть как-то предсказать неизвестную структуру по уже известной.
Если две последовательности по итогам такого выравнивания совпали в более чем 25% позиций, их трёхмерные структуры будут очень похожи — это эмпирическая закономерность. Если последовательности идентичны менее чем в 20% позиций, трёхмерные структуры будут сильно расходиться. Последовательности с совпадениями между 20 и 25% в смысле 3D-сходства лежат в некой «сумеречной зоне». Конечно, трёхмерную схожесть оценивают не на глаз, а с помощью параметра RMSD (Root Mean Square Deviation, среднеквадратичное отклонение), среднеквадратичной разности координат атомов трёхмерных структур. Мы накладываем две 3D-структуры друг на друга так, чтобы атомы как можно большего числа аминокислотных остатков этих двух полипептидных цепей оказались бы в (почти) одном и том же месте, то есть с почти одинаковыми координатами, или хотя бы в более или менее близком соседстве — и тогда координаты будут отличаться. Чем меньше (в среднем) эти отличия, то есть чем меньше RMSD, тем полнее совпадают пространственные структуры. Опыт показывает, что если последовательности идентичны на 30%, RMSD 3D-структур составляет около 1,4 ангстрема, при идентичности 20% — около 2 ангстрем, а при 15—10% идентичности RMSD становится больше 5—10 ангстрем и приближается к радиусу белка.
